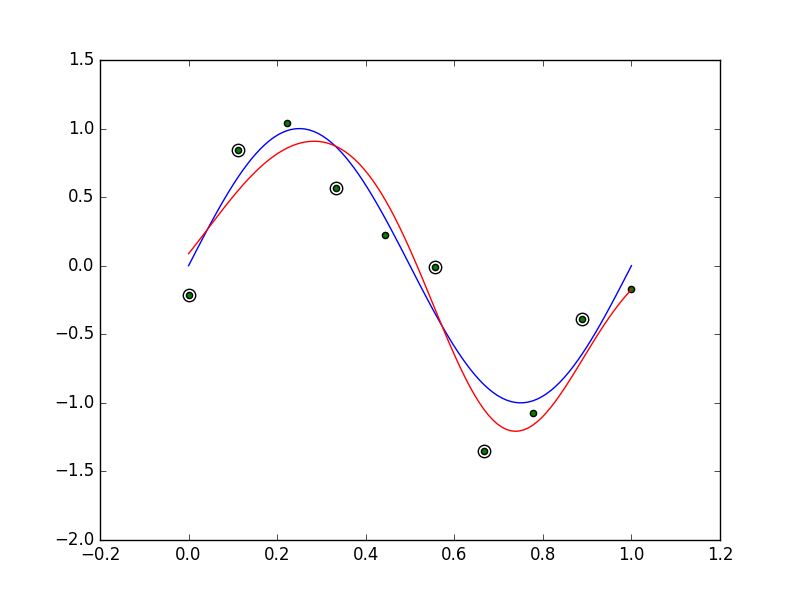
因果推論1 単純な選択バイアス問題
統計的因果推論の勉強

調査観察データの統計科学―因果推論・選択バイアス・データ融合 (シリーズ確率と情報の科学)
- 作者: 星野崇宏
- 出版社/メーカー: 岩波書店
- 発売日: 2009/07/29
- メディア: 単行本
- 購入: 29人 クリック: 285回
- この商品を含むブログ (25件) を見る
問題
- 大学の入試の結果y1と入学後の成績y2の関係を調べたい。
設定
- 真のモデル
- 入試の平均50、分散40
- 入学後の成績の平均50、分散40
- 2つのテストの相関0.8
- 正規分布に従うと仮定。
- [0, 100]となるように切り捨てる
- 選択バイアス
- 入試の合格点を60とする。
- 当然、入試の60点以下の人の入学後の成績は欠測となる。
を欠測のインディケータとする。この場合、合格のとき
, 不合格のとき
とする。
 - 赤い影になっている部分が欠測データ。
- 赤い影になっている部分が欠測データ。
回帰分析モデルを仮定する。
- モデル
- 最尤推定量
- つまり、
を用いた最小二乗法を行えばよい。
- このときの
の相関は、
parameters: [ 0.63938213 16.73959436] correlation: 0.820560498514
2変量正規分布に従うと仮定する。
- likelihood
- observed data likelihood
- [tex: p(y_1, y_2|\theta) = \prod{i, m_i=1}{p(y(i1), y(i2) | \theta)} \prod{i, m_i=0} p(y(i1) | \theta)]
- つまり、observed data likelihoodを最大化すれば良い。
mu1: 49.73650529 mu2: 48.54009208 sigma1: 10.18582171 sigma2: 7.93686404 rho: 0.820564
Python
import numpy as np import pandas as pd import matplotlib.pyplot as plt import math from scipy.stats import norm, multivariate_normal from scipy.optimize import minimize def negative_log_likelihood(y1, y2, m, m1, m2, s1, s2, rho): mean = np.array([m1, m2]) s12 = rho * s1 * s2 cov = np.array([[s1 * s1, s12], [s12, s2 * s2]]) v = 0.0 for yi1, yi2, mi in zip(y1, y2, m): if mi == 1: v += multivariate_normal.logpdf(np.array([yi1, yi2]), mean=mean, cov=cov) else: v += norm.logpdf(yi1, m1, s1) return -v if __name__ == '__main__': n = 1000 u1 = 50 u2 = 50 v1 = 100 v2 = 50 rho = 0.8 v12 = rho * math.sqrt(v1 * v2) mean = np.array([u1, u2]) cov = np.array([[v1, v12], [v12, v2]]) y = np.random.multivariate_normal(mean, cov, n) data = pd.DataFrame({'y1': y[:, 0], 'y2': y[:, 1]}) data['m'] = data.apply(lambda x: 1 if x['y1'] > 60 else 0, axis=1) data['y2'] = data.apply(lambda x: None if x['m'] == 0 else x['y2'], axis=1) pass_idx = data['m'] == 1 # using regression model coeff = np.polyfit(data[pass_idx]['y1'], data[pass_idx]['y2'], 1) c0 = coeff[0] var_y1 = np.std(data['y1']) ** 2 var_epsilon = np.std(data['y2'] - np.poly1d(coeff)(data['y1'])) ** 2 corr = c0 * var_y1 / (math.sqrt(var_y1) * math.sqrt(c0 ** 2 * var_y1 + var_epsilon)) print('parameters:', coeff) print('correlation:', corr) # using multivariate normal model. cons = ( {'type': 'ineq', 'fun': lambda theta: 0.9999 - theta[4]}, {'type': 'ineq', 'fun': lambda theta: theta[4] + 0.9999}, {'type': 'ineq', 'fun': lambda theta: theta[0:4] - 1}, {'type': 'ineq', 'fun': lambda theta: 100 - theta[0:4]}, ) theta0 = np.array([50, 60, 10, 30, 0.1]) theta = minimize(lambda theta: negative_log_likelihood(data['y1'], data['y2'], data['m'], theta[0], theta[1], theta[2], theta[3], theta[4]), theta0, constraints=cons, method="SLSQP") print(theta) axis_x = np.linspace(0, 100, 101) plt.scatter(y[:, 0], y[:, 1]) plt.fill_betweenx((-10, 110), 60, facecolor='r', alpha=0.2) plt.xlim(0, 100) plt.ylim(0, 100) plt.plot(axis_x, np.poly1d(coeff)(axis_x)) plt.xlabel('y1') plt.ylabel('y2') plt.show()
PRML 7. Sparse Kernel Machines (Regression with SVM and RVM)
- PRML本の7章でサポートベクターマシン等を使った回帰。
- 機械学習等は勉強中で、内容に誤りがある可能性が十分にあります。指摘していただけると嬉しいです

- 作者: C.M.ビショップ,元田浩,栗田多喜夫,樋口知之,松本裕治,村田昇
- 出版社/メーカー: 丸善出版
- 発売日: 2012/04/05
- メディア: 単行本(ソフトカバー)
- 購入: 6人 クリック: 33回
- この商品を含むブログ (18件) を見る
概要
- しきい値
から超えた距離
を最小化するように最適化を行う。
- svmではしきい値はgivenとするが、
-svmでは、
も最適化パラメータとする。このとき、
と
がトレードオフの関係になる。ただし、ラグランジュ未定乗数法の式変形の仮定で消える。
-svmは下記の最小化問題を解く。
- Relevance Vector Machine (RVM)はベイジアンなkernel method.
- 3章と同様に、事前分布のパラメータをiterativeに解く。
- SVMと比べ、予測時に使用する入力データの数が少なくなりやすい。
図7.8の再現
図7.9の再現
Python
- Python初心者なので、まだまだ汚いです。そのうち修正する。
import numpy as np import matplotlib.pyplot as plt from scipy.optimize import minimize import math class NuSvmRegression: def __init__(self, nu, c, sigma): self.nu = nu self.c = c self.sigma = sigma self.x = None self.t = None self.b = None self.d = None def kernel(self, xn, xm): return math.exp(- 0.5 * np.linalg.norm(xn - xm) ** 2 / (self.sigma ** 2)) def __call__(self, a): n = a.shape[0] // 2 a0, a1 = a[0:n], a[n:2 * n] d = (a0 - a1).flatten() k = np.array([self.kernel(xn, xm) for xn in self.x for xm in self.x]).reshape(n, n) v = - np.sum(d * self.t) for i in range(n): for j in range(n): v += 0.5 * d[i] * d[j] * k[i][j] return v def learn(self, x, t): self.x = x self.t = t n = x.shape[0] initial_a = np.random.uniform(0, 1, n * 2) cons = ({'type': 'ineq', 'fun': lambda a: a}, {'type': 'ineq', 'fun': lambda a: self.c / n - a}, {'type': 'ineq', 'fun': lambda a: self.nu * self.c - np.sum(a[0:n] + a[n:2 * n])}, {'type': 'eq', 'fun': lambda a: np.sum(a[0:n] - a[n:2 * n])}) res = minimize(self, initial_a, method='SLSQP', tol=1e-6, constraints=cons) a = res.x a0, a1 = a[0:n], a[n:2 * n] d = (a0 - a1) d = np.array([d0 if math.fabs(d0) > 1e-4 else 0 for d0 in d]).reshape(1, -1) k = np.array([self.kernel(xn, xm) for xn in self.x for xm in self.x]).reshape(n, n) self.b = np.average(t - np.dot(d, k)) self.d = d.flatten() return res.x def predict(self, x): n = self.t.shape[0] k = np.array([self.kernel(x, xm) for xm in self.x]) return np.sum(self.d * k) + self.b class RvmRegression: def __init__(self, sigma): self.sigma = sigma self.m = None self.mp = None self.cov = None self.alpha = None self.beta = None self.x = None def calculate_kernel(self, x): n = x.shape[0] k = [self.kernel(xn, xm) for xn in x for xm in x] k = np.array(k).reshape(n, n) k = np.hstack([k, np.ones(n).reshape(-1, 1)]) return k def kernel(self, xn, xm): return math.exp(- 0.5 * np.linalg.norm(xn - xm) ** 2 / (self.sigma ** 2)) @staticmethod def calculate_mu(beta, k, t, sigma): return beta * np.dot(sigma, np.dot(k.T, t)) @staticmethod def calculate_cov(a, beta, k): return np.linalg.inv(np.diag(a) + beta * np.dot(k.T, k)) @staticmethod def calculate_gamma(a, sigma): return 1 - a * np.diag(sigma) def learn(self, x, t, iteration=1): n = x.shape[0] a = np.random.uniform(10, 100, n + 1) beta = np.random.uniform(10, 100, 1)[0] k = self.calculate_kernel(x) for c in range(iteration): cov = self.calculate_cov(a, beta, k) mu = self.calculate_mu(beta, k, t, cov) gamma = self.calculate_gamma(a, cov) mu2 = np.array([a if a != 0 else 1e-16 for a in mu ** 2]) a = gamma / mu2 beta = (n - np.sum(gamma)) / (np.linalg.norm(t - np.dot(k, mu)) ** 2) self.m = mu self.cov = cov self.alpha = a self.beta = beta self.x = x self.mp = np.array([a if math.fabs(a) > 1e-4 else 0.0 for a in self.m]) def predict(self, x): k = np.array([self.kernel(x, xm) for xm in self.x]) k = np.append(k, 1) return np.inner(self.m, k) def predict2(self, x): k = np.array([self.kernel(x, xm) for xm in self.x]) k = np.append(k, 1) return np.inner(self.mp, k) def plot78(): np.random.seed(123) n = 10 x = np.linspace(0, 1, n) t = np.sin(2 * np.pi * x) + np.random.normal(0, 0.2, n) c = 10.0 * n sigma = 0.2 nu = 0.1 f = NuSvmRegression(nu, c, sigma) f.learn(x, t) idx = f.d != 0 axis_x = np.linspace(0, 1, 100) plt.clf() plt.plot(axis_x, np.sin(2 * np.pi * axis_x), 'b-') plt.plot(axis_x, [f.predict(x) for x in axis_x], 'r-') plt.scatter(x[idx], t[idx], marker='o', c='w', s=80) plt.scatter(x, t, marker='o', c='g') plt.savefig('img/f7.8.png') def plot79(): np.random.seed(123) n = 10 x = np.linspace(0, 1, n) t = np.sin(2 * np.pi * x) + np.random.normal(0, 0.2, n) sigma = 0.2 f = RvmRegression(sigma) f.learn(x, t, 400) idx = f.mp[0:n] != 0 axis_x = np.linspace(0, 1, 100) plt.clf() plt.plot(axis_x, np.sin(2 * np.pi * axis_x), 'b-') plt.plot(axis_x, [f.predict(x) for x in axis_x], 'r-') plt.scatter(x[idx], t[idx], marker='o', c='w', s=80) plt.scatter(x, t, marker='o', c='g') plt.savefig('img/f7.9.png') if __name__ == '__main__': plot78() plot79() print('hello')

- 作者: C.M.ビショップ,元田浩,栗田多喜夫,樋口知之,松本裕治,村田昇
- 出版社/メーカー: 丸善出版
- 発売日: 2012/04/05
- メディア: 単行本(ソフトカバー)
- 購入: 6人 クリック: 33回
- この商品を含むブログ (18件) を見る
PRML 7. Sparse Kernel Machines (Classification with SVM)
- PRML本の7章でサポートベクターマシンを使った分類アルゴリズム。
- 機械学習等は勉強中で、内容に誤りがある可能性が十分にあります。指摘していただけると嬉しいです

- 作者: C.M.ビショップ,元田浩,栗田多喜夫,樋口知之,松本裕治,村田昇
- 出版社/メーカー: 丸善出版
- 発売日: 2012/04/05
- メディア: 単行本(ソフトカバー)
- 購入: 6人 クリック: 33回
- この商品を含むブログ (18件) を見る
概要
- 分類の境界線から一番違い要素との距離を最大化するように最適化を行う。
- 2クラスの分類を扱うが、多クラスへの応用はストレートにできる。
- 最大化問題をラグランジュ未定乗数法を用いて双対問題を作り、双対問題を解く。
- 予測時に一部の学習データのみを用いる。それらをサポートベクターと呼ぶ。
- 式変形は単純なので、メモなし。
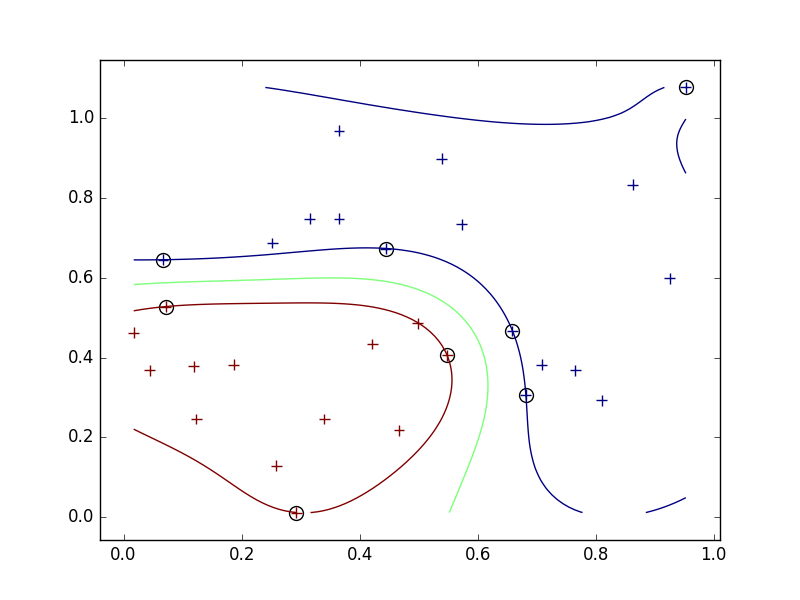
図7.2の再現
sigmaによる結果への影響について。
- 今回、Gaussianカーネルを使っているので、sigmaは各学習用データがその周りに与える影響の大きさを表している。
- 小さすぎるとサブセットの周りを囲むような境界線になってしまい、汎用性がさがる。
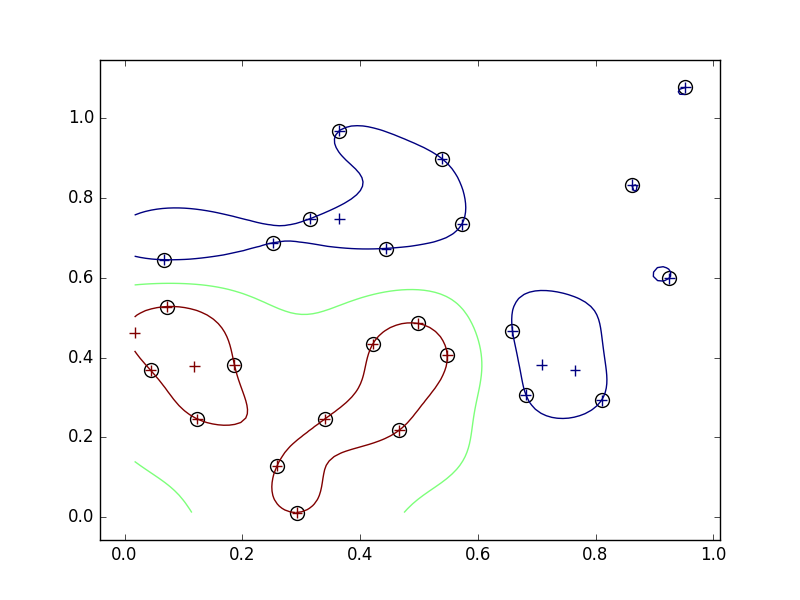
- 逆に大きくするとデータから遠いところが大雑把になりそう。
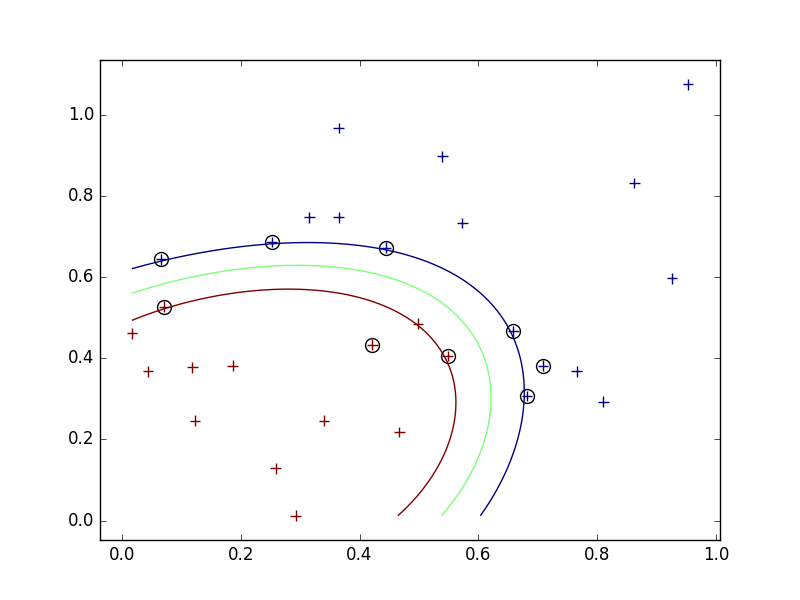
外れ値
- ナイーブに最大化問題を解くと、外れ値に対応できない。
- アルゴリズム上、すべての学習データを正しく分類する。汎用性が低くなる可能性がある。
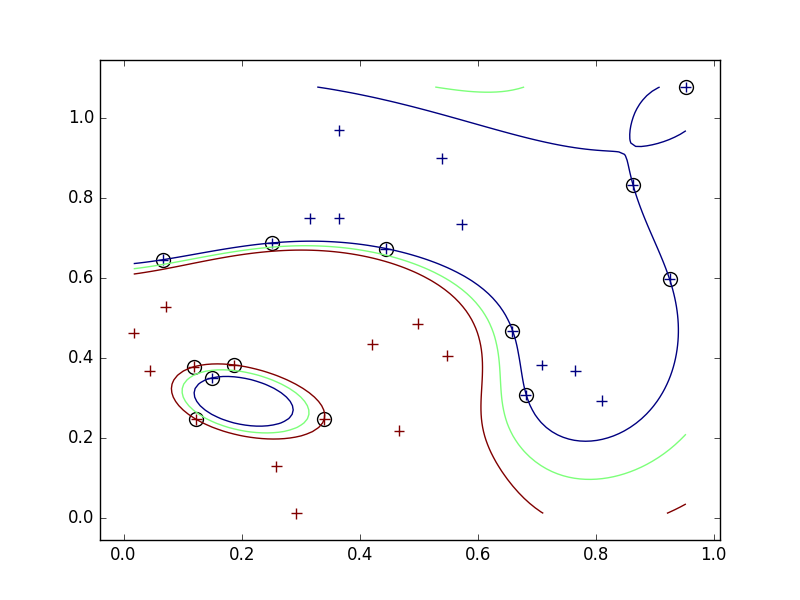
- そこで、スラック変数を導入して、誤って分類させることを許し、そのかわりにペナルティを与えるようにする。
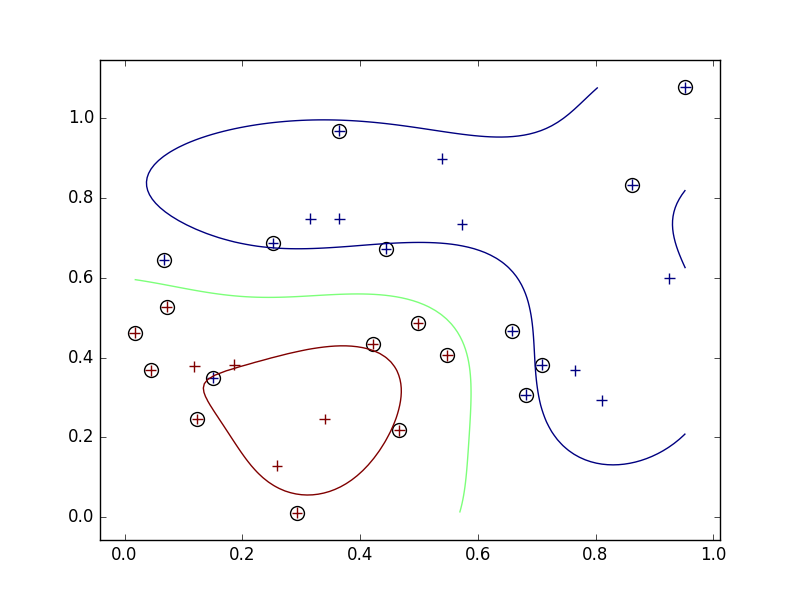
- スラック変数導入前は、左下の青い一点を囲むような境界ができていたが、導入後は左下の青い点は間違った分類(赤)になったままになった。
Python
import numpy as np import matplotlib.pyplot as plt from scipy.optimize import minimize import math class DualRepresentation: def __init__(self, x, t, sigma, c): self.x = x self.t = t.flatten() self.sigma = sigma self.a = None self.b = 0 self.c = c def kernel(self, xn, xm): return math.exp(- 0.5 * np.linalg.norm(xn - xm) ** 2 / (self.sigma ** 2)) def __call__(self, input): n = self.t.shape[0] a = np.array(input).flatten() k = np.array([self.kernel(xn, xm) for xn in self.x for xm in self.x]).reshape(n, n) v = np.sum(a) for i in range(n): for j in range(n): v -= 0.5 * a[i] * a[j] * self.t[i] * self.t[j] * k[i][j] return -v def set(self, a): n = self.t.shape[0] self.a = a idx = np.where(a != 0) ns = idx[0].shape[0] k = np.array([self.kernel(xn, xm) for xn in self.x for xm in self.x]).reshape(n, n) b = 0.0 for i in idx[0]: b += self.t[i] for j in idx[0]: b -= a[j] * self.t[j] * k[i][j] self.b = b / ns def learn(self): n = self.t.shape[0] a0 = np.random.uniform(0, 1, n) cons = ({'type': 'ineq', 'fun': lambda a: a}, {'type': 'eq', 'fun': lambda a: np.sum(self.t * a) - 1}, {'type': 'ineq', 'fun': lambda a: self.c - a}) res = minimize(self, a0, method='SLSQP', tol=1e-6, constraints=cons) a = np.array([x if x > 1e-12 else 0.0 for x in res.x]) self.set(a) return a def predict(self, x): n = self.t.shape[0] k = np.array([self.kernel(x, xm) for xm in self.x]) return np.sum(self.a * self.t * k) + self.b def plot72(x, t, sigma, c=1e+16, do_save=None): n = data.shape[0] f = DualRepresentation(data[:, 0:2], t, sigma, c) a = f.learn() n_plot = 100 axis_x0 = np.linspace(np.min(x[:, 0]), np.max(x[:, 0]), n_plot) axis_x1 = np.linspace(np.min(x[:, 1]), np.max(x[:, 1]), n_plot) boundary = np.array([f.predict(np.array([x0, x1])) for x0 in axis_x0 for x1 in axis_x1]).reshape(n_plot, n_plot) idx = np.where(a != 0.0) plt.clf() plt.scatter(x[idx, 0], x[idx, 1], c='w', marker='o', s=100) plt.scatter(x[:, 0], x[:, 1], c=t, marker='+', s=60) plt.contour(axis_x0, axis_x1, boundary.T, np.array([-1, 0, 1])) if do_save is None: plt.show() else: plt.savefig(do_save) if __name__ == '__main__': data = np.genfromtxt('dataset/classification_7_2.csv', delimiter=',').astype(np.float32) plot72(x=data[:, 0:2], t=data[:, 2], sigma=0.1, do_save='img/f7.2_0.1.png') plot72(x=data[:, 0:2], t=data[:, 2], sigma=0.2, do_save='img/f7.2_0.2.png') plot72(x=data[:, 0:2], t=data[:, 2], sigma=0.5, do_save='img/f7.2_0.5.png') data = np.vstack([data, np.array([[0.15, 0.35, -1]])]) plot72(x=data[:, 0:2], t=data[:, 2], sigma=0.2, do_save='img/f7.2_0.2_outlier.png') plot72(x=data[:, 0:2], t=data[:, 2], sigma=0.2, c=1, do_save='img/f7.2_0.2_outlier_slack.png')

- 作者: C.M.ビショップ,元田浩,栗田多喜夫,樋口知之,松本裕治,村田昇
- 出版社/メーカー: 丸善出版
- 発売日: 2012/04/05
- メディア: 単行本(ソフトカバー)
- 購入: 6人 クリック: 33回
- この商品を含むブログ (18件) を見る
2017年の目標
今年は去年より勉強に使える時間が増えそうなので、がっつり勉強したい。転職時に1ヶ月程度間を入れてじっくりと学べるかな。
(1) 機械学習を学ぶ
クオンツからデータアナリストに変わろうと思う。統計等は学部時代に勉強していたけど、機械学習はさらっとライブラリの使い方解説本を読んだ程度。プロとして最低限のレベルに達したい。
(2) 分析経験を積む
「いかに使うか」が重要なので、経験値を積みたい。業務で使用するだろうけど、幅広く色々分析したいので、ちょっと意識してデータを分析していく。
- 1ヶ月に1つ程度、データを分析して、その過程をブログにアップする。
(3) その他
教養がないので、世界史や生物学の本を読もうと思う。何がいいのかわからないけど、本やで適当に探そう。あと、インプットだけでなくアウトプットをしっかりと出していきたい。
- 1ヶ月に1冊読んで、感想をブログに書く。
- ブログを続ける
変化のある年に
10年近く勉強してきたクオンツ業界から離れることを決意したので、節目の年になる。新しい分野でしっかりと結果を出していきたい。
Jenkinsメモ
github, jenkins, aws
git
submodule
- Reference
- Git submodule の基礎
- git submoduleを今風な感じで削除する
jenkins
plugin
- Reference
- How to install a plugin in Jenkins manually?
- MSBuild Plugin
- After installation, there is setting form in "Global Tool Configuration".
job
- Reference
- Hudson/JenkinsでVisual Studioプロジェクトのビルドをする
- JenkinsとMSBuild PluginでVisual Studioのプロジェクトをビルドしてみたよ
- example
- MSBuild sample.sln /t:Build /p:VisualStudioVersion=14;Configuration=Debug;Platform=x86
with google test
job flow
使い方のメモ。
githubからコードを入手
https://github.com/Excel-DNA
ExcelDna\Sourceにあるソリューションを開いてでビルドする。
ExcelDna\Build\build.batを実行。
新しいC#プロジェクトを作成。
ExcelDna\DistributionにあるExcelDna.Integrationを参照に追加。
namespace Excel { public class Export { public static double Add(double x, double y) { return x + y; } } }
ExcelDna.dnaを編集
<DnaLibrary RuntimeVersion="v4.0"> <ExternalLibrary Path="Excel.dll" /> </DnaLibrary>
ExcelDna.xllを開いて、新しいbookを作成し、Addを呼んでみる。
動いた!
Rangeを返す。
public static object[] ReturnArray(int n) { object[] array = new object[n]; for (int i = 0; i < n; ++i) { array[i] = i; } return array; } public static object[,] ReturnMatrix(int n) { object[,] array = new object[n, n]; for (int i = 0; i < n; ++i) { for (int j = 0; j < n; ++j) { array[i, j] = i + j; } } return array; }
Rangeを受け取る
public static string UpdateRange(object[,] range) { for (int i = 0; i < range.GetLength(0); ++i) { for (int j = 0; j < range.GetLength(1); ++j) { range[i, j] = i + j; } } return "success!!"; }
ん?? 更新できない・・・
できないことはないようだが、推奨されていないみたい。
https://groups.google.com/forum/#!topic/exceldna/SCPBnuw7Cl8
tupleを展開して、関数の引数にする。
したいこと。
int main() { auto x = std::make_tuple(1, 2.0, "three"); auto f = [](auto a, auto b, auto c) { std::cout << typeid(a).name() << std::endl; std::cout << typeid(b).name() << std::endl; std::cout << typeid(c).name() << std::endl; }; apply(f, x); return 0; }
上記のようにapplyに関数とtupleを渡した時に、下記のように関数を評価したい。
f(1, 2.0, "three");
variadic templateを再帰的に呼び出すことによって、tupleの中身を順次Args&... argsに貯める。最後に関数fに渡す。
template <int N> struct Expand { template <typename F, typename Tuple, typename... Args> static void apply(F& f, Tuple& t, Args&... args) { Expand<N - 1>::apply(f, t, std::get<N - 1>(t), args...); } }; template <> struct Expand<0> { template <typename F, typename Tuple, typename... Args> static void apply(F& f, Tuple& t, Args&... args) { f(args...); } }; template <typename F, typename Tuple> void apply(F& f, Tuple& t) { Expand<std::tuple_size<Tuple>::value>::apply(f, t); }
実行イメージ
Expand<3>::apply(f, x)
Expand<2>::apply(f, x, "three")
Expand<1>::apply(f, x, 2.0, "three")
Expand<0>::apply(f, x, 1, 2.0, "three")
f(1, 2.0, "three")